蛋白翻译后修饰(Post-Translational Modification, PTM),例如磷酸化,甲基化,乙基化,糖基化等,是蛋白质构象及信号传导的关键调节剂。对于翻译后修饰蛋白,冷冻电镜无法研究其全谱动态,Deepmind公司开发的Alphafold2是一种基于神经网络的模型算法对蛋白质三维结构进行准确计算预测的工具,它的一般用法也不能准确捕捉各种PTM对蛋白折叠及蛋白间相互作用的影响。
生物质谱是蛋白一级结构测序以及蛋白翻译后修饰类型以及位点鉴定的最佳工具。化学生物学平台与香港科技大学唐少军组合作,提出了预测翻译后修饰刺突蛋白三维结构的新方法。在蛋白质一级结构上用计算机进行氨基酸取代的方法以模拟PTM位点改变的物理化学性质,然后使用Alphafold2重新构建蛋白结构。以新冠病毒刺突蛋白为例,用生物质谱数据检测得到刺突蛋白上的几种翻译后修饰位点(图1),再通过用酸性氨基酸替换磷酸化氨基酸,用初级氨基酸序列进行Alphafold2计算机预测(图2)。研究表明与冷冻电镜实验相比,刺突蛋白受体结合区域(RBD)结构域发生了显着变化。项目进一步分析了SARS-CoV-2刺突RBD与人ACE2的结合亲和力和分子间相互作用,以证明PTM可以显著影响ACE2的相互作用位点。

图1:SARS-CoV-2 S刺突蛋白5个主要修饰位点的87个PTM位点分布
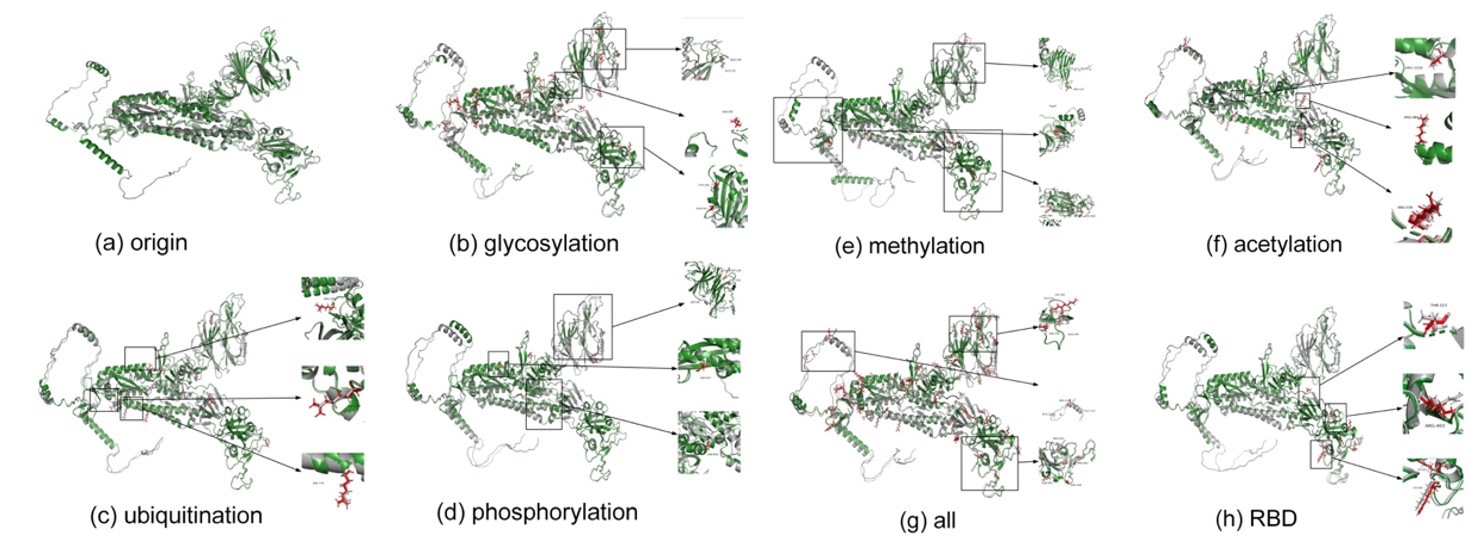
图2:重建PTM修饰的刺突蛋白三维结构
合作研究成果以“ SARS-CoV-2 Spike Protein Post-Translational Modification Landscape and Its Impact on Protein Structure and Function via Computational Prediction” 为题发表在Research上,并被岚翰生命科学等报道和转载。
分析中心化学生物学平台将相继推出相关新方法的科研服务。
